DATE2024.10.22 #Press Releases
トランスオミクスネットワークを構築・解析するwebアプリ「iTraNet」の開発
—— 生体システムの包括的理解に向けて——
発表のポイント
- mRNAやタンパク、代謝物などの多階層にまたがる相互作用を繋いだトランスオミクスネットワークを簡便に構築・解析できるwebアプリ「iTraNet」を開発した。
- 手間がかかると同時にデータベースのバージョンの違いなどにより再現性が低かった多階層オミクスデータの統合解析を、本webアプリにより簡便かつ再現性よく行えるようになった。
- オミクスデータ解析の効率化、そして、生命システムの包括的理解への貢献が期待される。
mRNAやタンパク、代謝物などの関係性を推定、解析するwebアプリ「iTraNet」
発表概要
東京大学大学院医学系研究科の杉本光大学院生と、東京大学大学院理学系研究科附属遺伝子実験施設の守田啓悟助教、大学院理学系研究科の黒田真也教授らによる研究グループは、RNA-seqや質量分析などを用いて測定した多種類のオミクスデータから、トランスオミクスネットワーク(注1)を構築・解析できるwebアプリ「iTraNet」を開発しました。(図1)
さらに、iTraNetを用いることで、野生型マウスでは糖摂取後にハブ分子(注2)がまとまって変動する一方、肥満モデルマウスではそれらのハブ分子の変動が消失することを観測しました。このwebアプリにより、オミクスデータ解析の効率化と再現性の向上、そして、生体ネットワークの包括的理解に役立つことが期待されます。
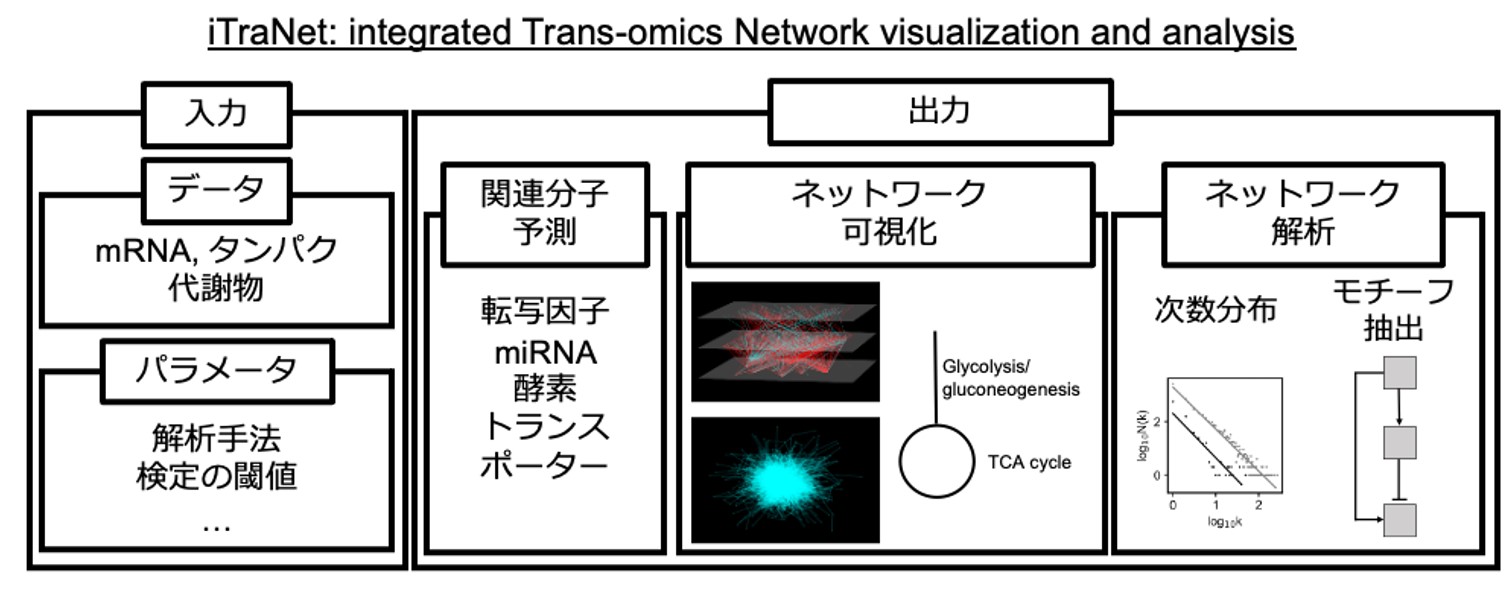 図1:iTraNetの概略
図1:iTraNetの概略
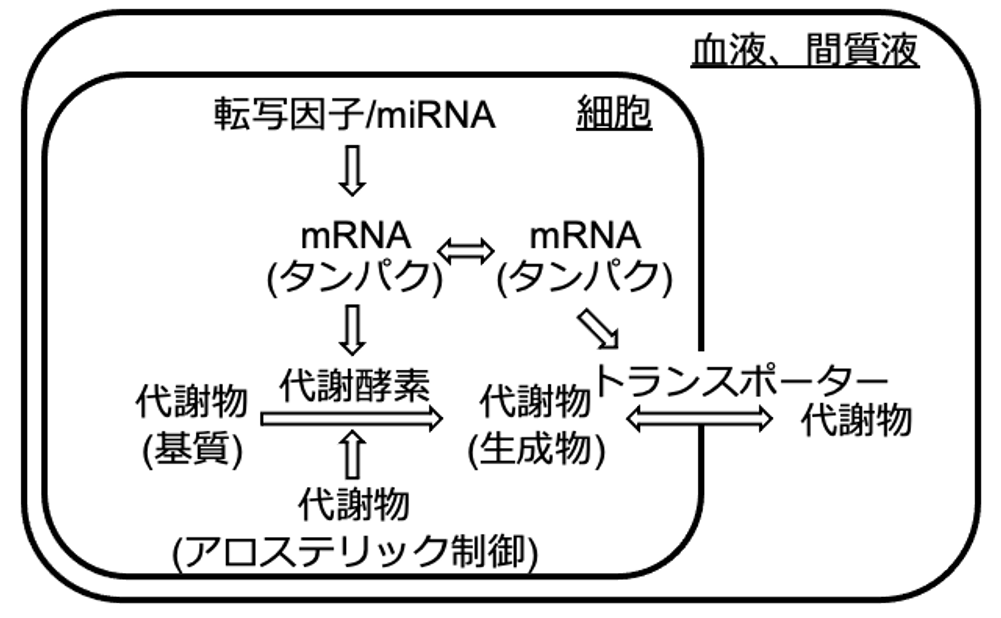
iTraNetは、ユーザーがアップロードしたトランスクリプトーム、プロテオーム、メタボロームなどのオミクスデータに関連する転写因子やマイクロRNA、酵素、トランスポーターなどを推定し、それらの関係性を繋いだネットワーク図を構築・可視化する。そして、そのネットワークの構造についての解析も行う。
発表内容
生命システムの包括的理解にむけて、mRNAやタンパク、代謝物などのさまざまな分子間の相互作用を包括的に解析することが必要とされています。RNA-seqや質量分析の発展によりmRNAやタンパク、代謝物を網羅的に計測することが可能になってきましたが、それら多種類のオミクスデータを統合して解析することは手間であると同時に、データベースのバージョンの違いなどにより再現性が低いことが問題点として挙げられていました。この度、本研究チームはmRNAやタンパク、代謝物などの多階層にまたがる相互作用を繋いだトランスオミクスネットワークを簡便かつ再現性よく構築・解析できるwebアプリ「iTraNet」を開発しました。
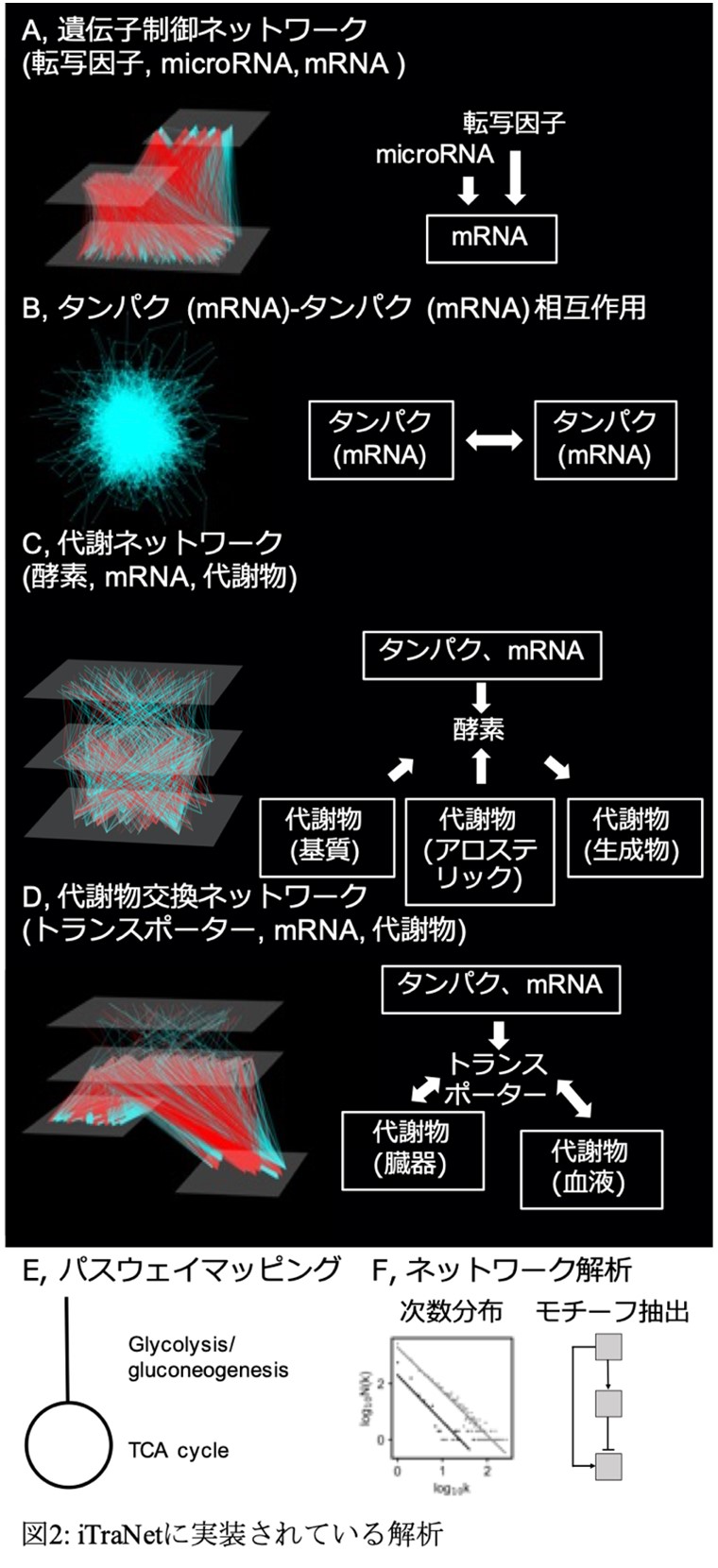
iTraNetはユーザーがアップロードしたトランスクリプトーム、プロテオーム、メタボロームなどのオミクスデータをもとに、遺伝子制御ネットワーク(A)、タンパク-タンパク相互作用(B)、代謝ネットワーク(C)、代謝物交換ネットワーク(D)の4種類のネットワークを構築、可視化します。特に、アロステリック制御(注3)や細胞内外の代謝物交換など、従来のツールでは考慮されていなかった制御メカニズムも含めた解析が可能な点がiTraNetの特徴の一つです。さらに、iTraNetは、よく知られた配置を用いて各代謝経路を表示することができます(E)。これにより、複雑な生物学的ネットワークをより直感的に理解することが可能になります。さらに、次数分布やモチーフ抽出などのネットワーク解析の結果も出力されます(F)。これにより、生命システム全体の特性を理解するために重要な情報が得られることが期待されます。(図2)
iTraNetのケーススタディとして、野生型マウスと肥満モデルマウスにおけるグルコース投与後の代謝状態の比較を行いました。この解析により、野生型マウスではトランスオミクスネットワークのハブ分子がグルコース投与に応答する傾向があるのに対し、肥満モデルマウスではこの傾向が失われていることが明らかになりました。
iTraNetは、オミクス解析の効率化と再現性の向上、そして、生命システムの包括的理解に貢献することが期待されます。本ツールは、https://itranet.streamlit.app/から利用可能です。また、ソースコードはGitHubリポジトリ(https://github.com/HikaruSugimoto/Transomics_iTraNet)で公開されており、研究者が自由に改変して使用することができます。
〇関連情報:
「肥満による摂食時の代謝異常をマウス肝臓のトランスオミクスネットワーク解析により解明」(2024/2/27)
「肝臓-筋肉の臓器連関代謝サイクルの肥満による制御異常の解明」(2021/3/10)
関連リンク:科学技術振興機構(JST)
論文情報
-
雑誌名 Bioinformatics Advances 論文タイトル iTraNet: A Web-Based Platform for integrated Trans-Omics Network Visualization and Analysis著者 Hikaru Sugimoto, Keigo Morita, Dongzi Li, Yunfan Bai, Matthias Mattanovich, Shinya Kuroda*
(*責任著者)DOI番号 10.1093/bioadv/vbae141
研究助成
本研究は、科学技術振興機構(JST)における戦略的創造研究推進事業 CREST「多細胞間での時空間的相互作用の理解を目指した定量的解析基盤の創出」研究領域 研究課題名「時空間トランスオミクスを用いた多細胞・臓器連関代謝制御の解明」(課題番号:JPMJCR2123 研究代表者:黒田真也)、科研費学術変革領域研究(A)「冬眠・休眠のトランスオミクス解析(課題番号:23H04946)」、「冬眠生物学2.0 総括班(課題番号:23H04939)」、武田科学振興財団医学部博士課程奨学助成などの支援により実施されました。
用語解説
注1 トランスオミクスネットワーク
複数の階層(遺伝子、タンパク質、代謝物など)にまたがる生体内分子の関係を繋いだネットワーク。↑
注2 ハブ分子
ネットワーク内で多くの他の分子と相互作用を持つ分子。ネットワーク全体の機能や挙動に大きな影響を与える傾向にある。↑
注3 アロステリック制御
酵素の活性部位以外の部位に代謝物などが結合して、酵素の活性が調節される機構。↑


