DATE2023.08.01 #Press Releases
シングルセルRNA-seqデータから細胞種を同定する深層学習
――新しいアーキテクチャscDeepInsight法を開発――
ジア シャンルー(大学院新領域創成科学研究科 修士課程)
角田 達彦(生物科学専攻 教授/大学院新領域創成科学研究科(兼担)/理化学研究所 チームリーダー)
アルテム ルイセンコ(生物科学専攻 助教/理化学研究所 客員研究員)
アロック シャルマ(理化学研究所 専任研究員/グリフィス大学 教授/大学院理学系研究科 客員共同研究員)
キース ボロエヴィッチ(理化学研究所 テクニカルスタッフI)
発表のポイント
- シングルセルRNA-seqデータからもとの細胞種を同定する、深層学習を用いた新しい手法scDeepInsightを提案しました。
- 1細胞ごとの発現データを前処理後に画像化することにより、深層学習が持つ画像分類や特徴抽出の利点を最大限に活用できます。それにより、細胞種を高い精度で同定をしたり、希少あるいは未知の細胞種を検出したりすることができます。
- 本手法を用いることにより、生体内や疾患における細胞制御のメカニズムの解明に貢献することが期待されます。
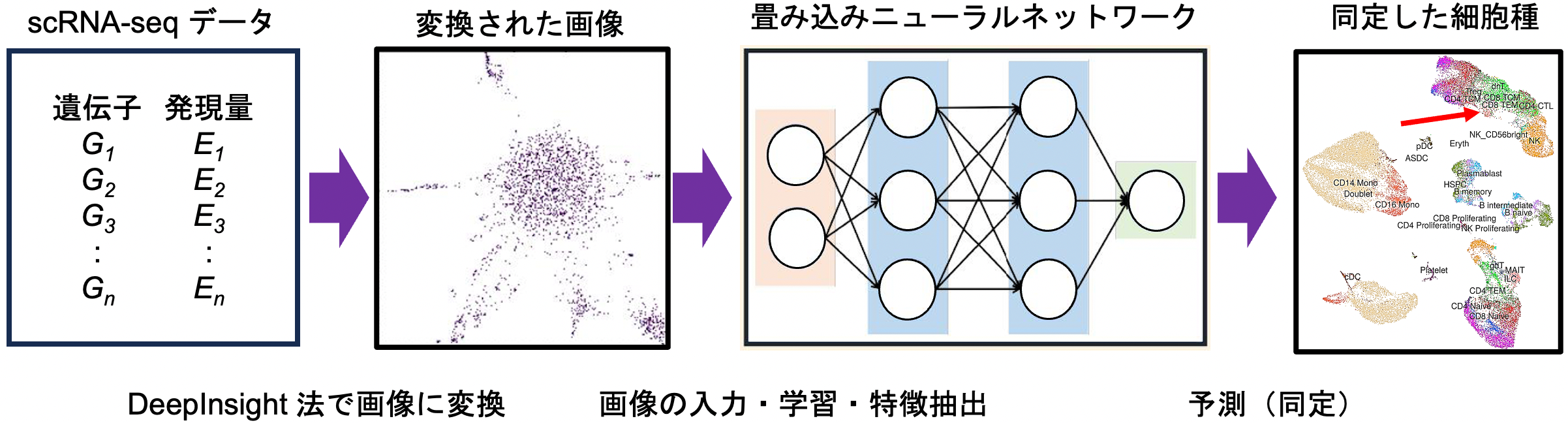
提案手法scDeepInsightの概要図
発表概要
東京大学大学院新領域創成科学研究科のジア シャンルー大学院生、理化学研究所生命医科学研究センターのアロック シャルマ専任研究員、東京大学大学院理学系研究科のアルテム ルイセンコ助教、角田達彦教授らによる研究グループは、シングルセルRNA-seq(scRNA-seq;注1)のデータからもとの細胞種を同定する、深層学習(注2)を用いた新しい手法scDeepInsightを提案しました。細胞種を同定することは、scRNA-seqデータの解析によって細胞集団の不均一性を研究するための鍵となります。この手法では、scRNA-seqの非画像のデータを、私たちが独自に提案してきたDeepInsight法(注3)を使って画像に変換することにより、画像分類や特徴抽出能力が高い畳み込みニューラルネットワーク(CNN;注4)を活用することができます。その結果、scDeepInsightは他の最先端の手法に比べてはるかに高い精度で細胞種を同定することができました。本提案手法により、将来的に、scRNA-seqデータを用いた生体内や疾患における細胞制御のメカニズムの解明に関わる研究に広く貢献することが期待できます。
発表内容
〈研究の背景〉
近年急速に発展してきたシングルセルRNA-seq(scRNA-seq)の技術により、1細胞レベルで網羅的に遺伝子発現を観察できるようになり、多種多様な組織とその中のさまざまな種類の細胞の構成を詳細に調べることができるようになりました。このscRNA-seqデータを解析する過程で、正確に細胞種を同定する必要があります。その同定は、計算機によるある程度の精度での推定の後の専門家による経験的な分析に基づいた手動での同定に頼ることが多かったのですが、最近は膨大なscRNA-seqデータが測定されるようになったため、専門家による同定ではスピードが追いつかないという問題が出てきました。さらに手作業による同定は時間と手間がかかるだけでなく、結果が主観的なものになる可能性もあります。これらをふまえ、計算機による推定の精度を上げ同定を自動化することが重要な研究課題となっています。
ところで、scRNA-seqデータは、高次元で、観測値が欠損している箇所が非常に多く、かつ背景に複雑なメカニズムがあることが多いという特性があります。このような場合、機械学習を用いることが適しています。参照データセットによって異なる細胞種の複数の遺伝子の発現パターンを学習しておけば、対象とする新しいデータの細胞種を同定することができます。特に深層学習はデータに潜む非常に抽象化された構造を学習することが可能です。本研究グループは、このような深層学習の特長と、細かく細胞種が注釈づけされた参照データセットが利用できることに着目して本研究を進めました。
〈研究の内容〉
本研究では、scRNA-seqデータから元となる細胞種を正確に同定するために、深層学習を用いた新しい手法scDeepInsight法を提案しました(図1)。本手法は参照データセットで学習させておいたモデルに基づき、対象とする未知のデータに対して、そのデータの元となった細胞種を同定することができます。最初のステップではデータの標準化などの前処理を行います。次に、私たちが独自に開発してきたDeepInsight法を用い、scRNA-seqデータを対応する画像に変換します。この方法をまず参照データセットに適用し、生成した画像を使ってCNNを学習させます。こうして構築した予測モデルを、今度は対象とするデータに対して細胞種を同定するために用います。scDeepInsightによって正確な細胞種が同定できる理由は、scRNA-seqデータを変換して画像化することによって、CNNの強みである特徴抽出と分類を活用できるからです。また、学習過程で抽出された特徴を、本研究グループが独自に開発してきたDeepFeature法(注5)を使って分析することにより、細胞種の性質を決めるためのマーカー遺伝子を見出すこともできます。さらに、参照データセットには含まれない、新しい細胞種を検出することも可能です。これにより、例えば病気にのみ現れる細胞種を抽出することもできます。
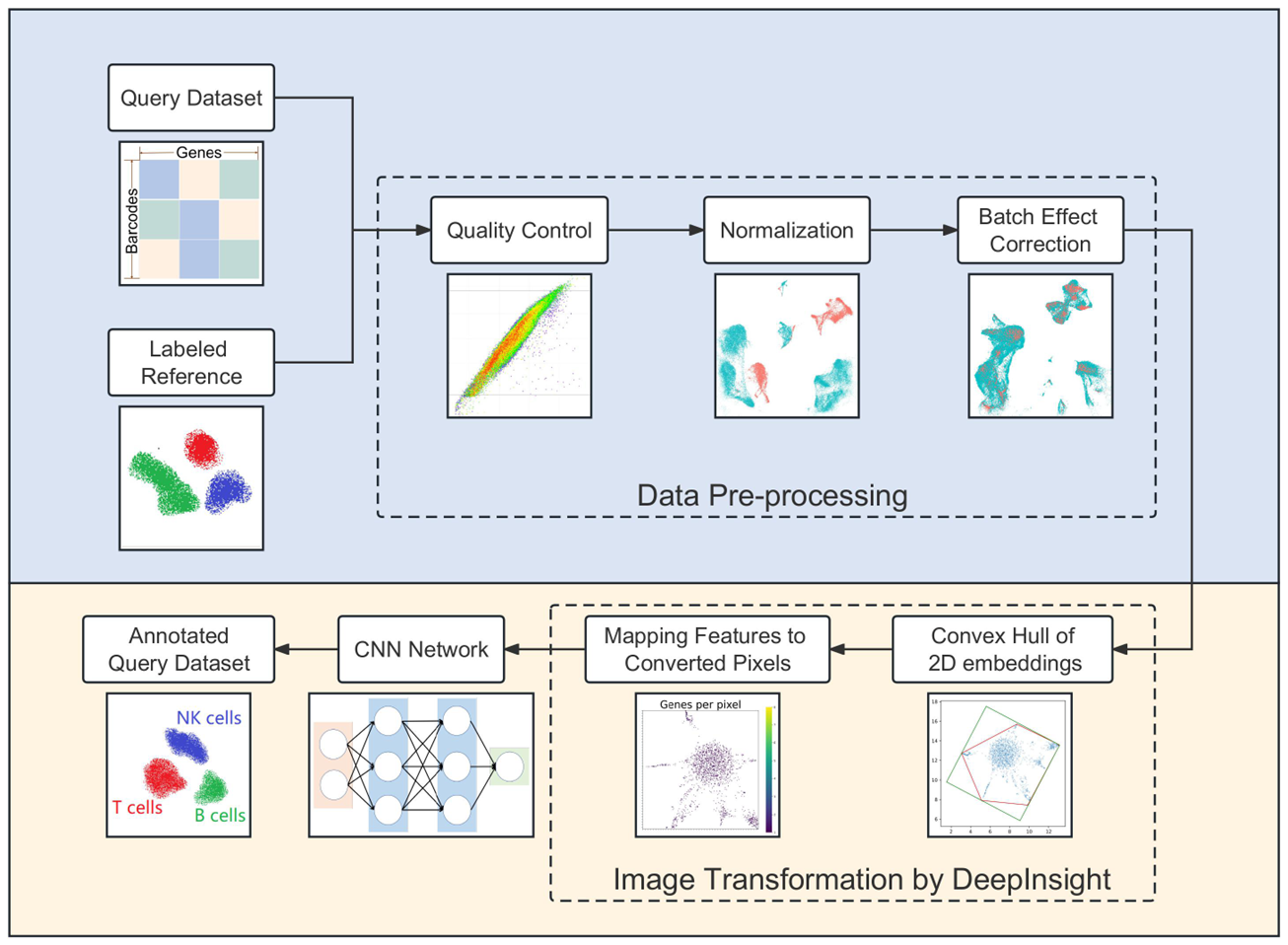
図1:scDeepInsightのパイプライン
scRNA-seqデータに対し、品質管理、正規化、バッチ効果の補正などの前処理をします。次に、1細胞の発現データを、対応する2次元画像に変換します。その後、参照データセットを使ってCNNを学習させます。CNNの学習が完了したら、それを新たなscRNA-seqデータの細胞種を同定するために使います。
今回提案したscDeepInsight法の性能を評価するため、末梢血単核細胞(PBMC)のscRNA-seqデータを用いました。PBMCは医科学研究で使われることが大変多く、解析する価値が非常に高いものです。しかし、免疫系の細胞などの互いに似つつも異なる細胞種・亜種が複数混ざっており、それらの組成比も非常にアンバランスなため、PBMC細胞を正しく同定することは困難な課題でした。本研究では、scDeepInsightの学習のための参照データセットとして、実験と専門家によってラベル付けされたPBMCデータセットを使用しました。この参照データセットには、160,000以上の細胞と31種類の細胞種が含まれています。そして新たなPBMCのテストデータセットを用いて細胞種を同定した結果、scDeepInsightの精度は、よく使われる6つの細胞種同定手法を7%以上、上回りました(図2)。
これに加えて、テストデータセットでの同定結果は元の細胞種のラベルと一致するだけでなく、さらにいくつかの細かい細胞種に分けることもできました(図3)。すなわち、これまでの手法に比べ、本手法は精度が高いだけでなく、類似しているが異なる細胞亜種の細かな違いを検出することが可能であることも分かりました。
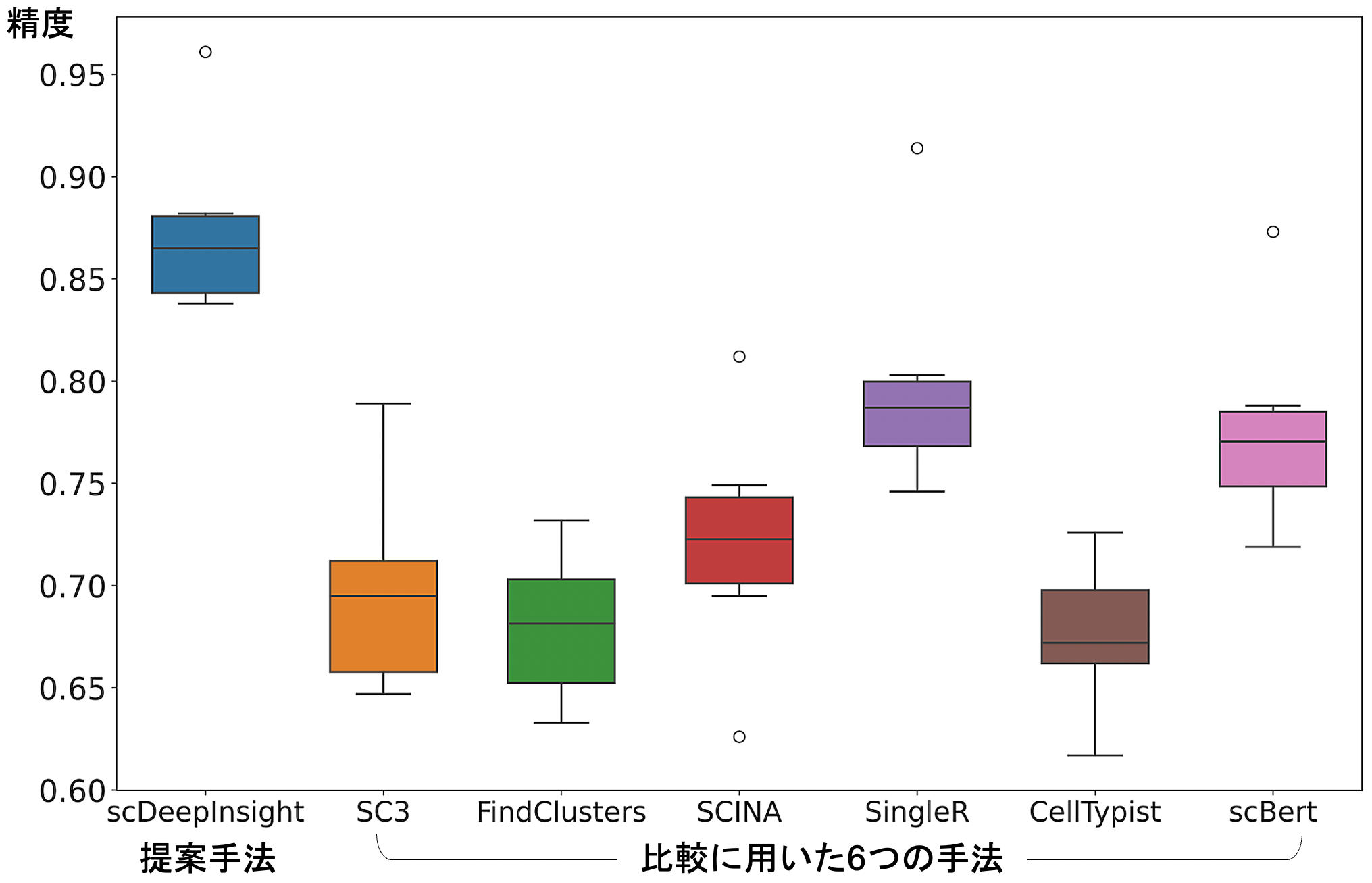
図2:提案手法scDeepInsight(左端)と他の6つの手法との精度の比較
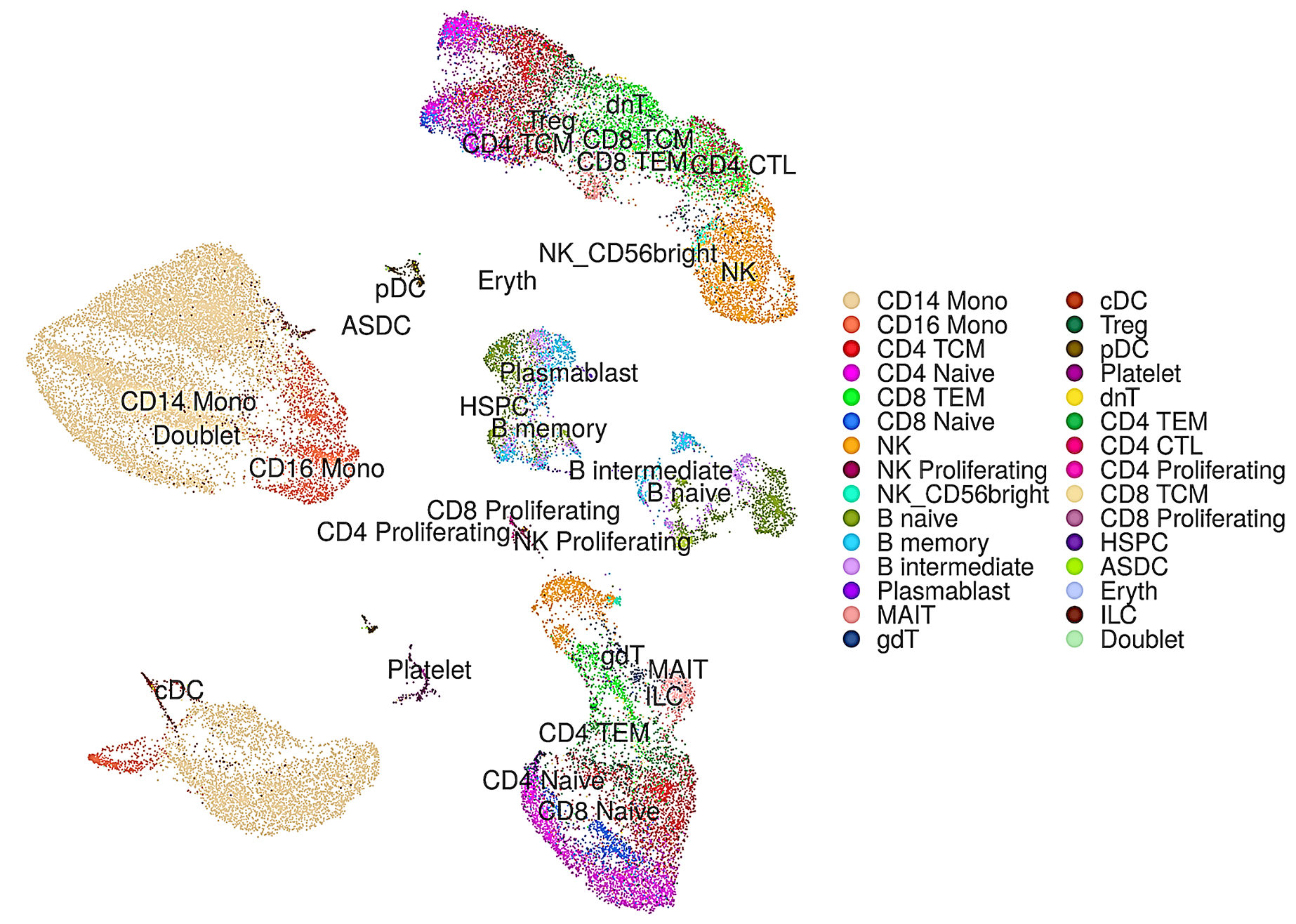
図3:scDeepInsightによって予測した細胞種ごとに色分けした結果
また、本提案手法scDeepInsightの大事な特長として、希少な、あるいは未知の細胞種を検出できることがあげられます。そのような細胞種が検出できれば、疾患の発症過程の理解や新しい治療法の開発につながります。例えば、新しい免疫細胞の亜種を検出することによって、新しい治療標的を発見したり、免疫系がウイルス感染にどのように反応するかなどの複雑な生体システムをよく理解したりすることにつながる可能性があります。しかし、解析対象とするデータに対して、参照データセットに含まれる既知の細胞種のどれかに分類するだけにとどめてしまうと、希少な細胞種や未知の細胞種が無視されてしまいます。この問題に対し、scDeepInsightでは、各細胞に対してそれぞれの細胞種として分類される確率を求めることにより、全ての細胞種に対してその確率が低ければ、サンプルが参照データセットの既知の細胞種のいずれにも類似しないものとして検出できます。このような場合、これらの細胞は未知の細胞種であると推定することができます。この能力を実証するために、COVID-19感染者の細胞を含む2つのデータセットで性能を評価しました。これらのデータセットには、一部の好中球、活性化ナチュラルキラー細胞、活性化CD4陽性T細胞、活性化CD8陽性T細胞が含まれており、これらは全て健康な人から得られた参照PBMCデータセットには存在しません。感染者から採取した細胞のデータの中で、既知細胞腫に分類される確率が1%以下のものを抽出すると、参照データセットに含まれない好中球などを未知の細胞種として弁別することができました。
そして、本研究グループが以前提案したDeepFeature法によって、学習済みのCNNの中味を解析することにより、CNNが予測する際に重要視する特徴を抽出することができます。今回の細胞種同定でも、複数の細胞種間で異なる量をもつ特徴を分析することができました。DeepFeatureをscRNA-seqデータセットに適用した結果見出された遺伝子には、その細胞種に対応するマーカー遺伝子が含まれていることが分かりました。
〈今後の展望〉
今後、異なる組織から得られた多くの参照データを統合し、より多くの細胞種を網羅する同定モデルを構築する予定です。また、他のオミクスデータも併せて解析することにより、細胞種同定の精度をさらに高めていきます。このような方法が進展すれば、がんや慢性疾患などのさまざまな病気の細胞制御のメカニズムに対する理解がますます深まると期待されます。
〈関連のプレスリリース〉
「人工知能でゲノミクスを」(2019/08/06)- DeepInsight法
https://www.s.u-tokyo.ac.jp/ja/press/2019/6493/
「人工知能はゲノミクスで何を見つめるのか?」(2021/08/19)- DeepFeature法
https://www.s.u-tokyo.ac.jp/ja/press/2021/7509/
「マルチオミクスと深層学習による抗がん剤奏効予測」(2023/02/14)- DeepInsight-3D法
(理化学研究所生命医科学研究センターニュース)
https://www.ims.riken.jp/2023/02/004489.php
論文情報
-
雑誌名 Briefings in Bioinformatics 論文タイトル scDeepInsight: a supervised cell-type identification method for scRNA-seq data with deep learning著者 Shangru Jia, Artem Lysenko, Keith A Boroevich, Alok Sharma*, Tatsuhiko Tsunoda*DOI番号
研究助成
本研究は、科研費「基盤研究B(課題番号:JP20H03240)」とJST CREST JPMJCR2231の支援により実施されました。
用語解説
注1 シングルセルRNA-seq(single-cell RNA sequencing; scRNA-seq)
次世代シークエンサーによって、1細胞ごとに、網羅的な遺伝子セットのmRNAの量などを求める方法。↑
注2 深層学習
深層学習は多層のニューラルネットワーク(ディープニューラルネットワーク)による機械学習手法。ディープラーニング。データを入力する入力層、演算をしながら情報を受け継いでいく複数の中間層、そして判別結果を出力する出力層からなる。↑
注3 DeepInsight法
ゲノミクスデータなどの非画像データを画像データに変換して深層学習で扱えるようにする、角田教授らが2019年に提案した独自の方法。↑
注4 畳み込みニューラルネットワーク(CNN)
特に画像の分類や識別で高い性能を発揮するディープラーニングの一つ。あらかじめ与えられていた画像データから画像の特徴量を直接抽出し、ネットワークを学習する。CNNはConvolutional Neural Networkの略。↑
注5 DeepFeature法
DeepInsight法でゲノミクスデータなどの非画像データを画像化し学習させたとき、深層学習が中で何を重視しているかを生物医学的に解釈可能にし、重要な遺伝子などの特徴を抽出する、 角田教授らが2021年に提案した独自の方法。 ↑


