DATE2023.01.06 #Press Releases
リボソーム衝突に依存したmRNAの内部切断の仕組み
東京大学医科学研究所
東京大学大学院新領域創成科学研究科
東京大学大学院理学系研究科
発表概要
リボソームは、mRNA上の遺伝暗号を解読しタンパク質の合成機能を担います。翻訳中のリボソームの停滞はタンパク質合成の異常を示す危険警告であり、停滞を引き起こしたmRNAと合成途中のペプチド鎖は、ユビキチン修飾を介して翻訳品質管理機構により速やかに分解されます。mRNAの切断酵素としてCue2が同定されていましたが、Cue2がどのようにユビキチン修飾を認識し、mRNAの切断部位を決定する仕組みは不明でした。
今回、東京大学医科学研究所RNA制御学分野/大学院新領域創成科学研究科メディカル情報生命学専攻/大学院理学系研究科生物科学専攻の稲田利文教授と東京大学大学院薬学系研究科の友松翔太大学院生、東北大学大学院薬学研究科の渡邊敦也大学院生は、エンドヌクレアーゼCue2のユビキチン結合活性が衝突したリボソームの5’側の外側でのmRNA切断に必要であることを明らかにしました。また、衝突したリボソームの間でのmRNA切断に必須な領域を一アミノ酸残基レベルで明らかにしました。
翻訳品質管理機構におけるリボソーム結合分子Mbf1の機能についても解析を行い、Mbf1がリボソーム停滞によって誘導される品質管理に寄与していることを明らかにし、その機能が停滞配列の種類に応じて異なることを明らかにしました。本成果はmRNA分解機構の理解につながるとともに、mRNAワクチンの開発の発展につながることが期待されます。本研究は、日本医療研究開発機構(AMED-CREST 課題番号:JP20gm1110010、研究代表者:稲田利文)、日本学術振興会科学研究費助成事業(課題番号:JP21H05277, JP19H05281, JP22H00401、稲田利文;21H00267, 21H05710, 22H02606、松尾芳隆)、 科学技術振興機構(JST)さきがけ(課題番号:JPMJPR21EE、研究代表者:松尾芳隆)などの支援を受けて行われました。
本研究成果は、米国科学誌「Nucleic Acids Research」(12月30日付けオンライン版)に公表されました。
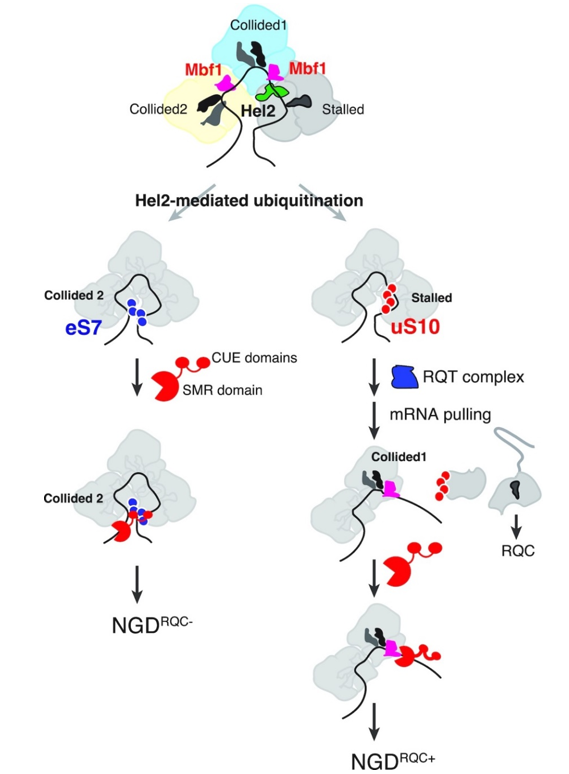
図:内在性の停滞配列によって誘発されるNGDの2つのモードの基質のCue2認識のモデル。Cue2のN末端領域に存在するユビキチン結合活性が、衝突したリボソームの上流でのmRNA切断に必須である。
NGDRQC-のモデル(左):Hel2は、SDD1 mRNA上に形成されたトリソーム内の衝突リボソームのeS7(青丸で表示)上にK63結合ポリユビキチン鎖を形成する。Cue2は、2つのCUEドメイン、CUE-D1および CUE-D2で eS7上のK63 結合ポリユビキチン鎖に結合し、衝突するリボソーム2の上流でmRNAを切断する。
NGDRQC+ のモデル(右):Hel2は主要なリボソームでuS10をユビキチン化し、RQT複合体はポリユビキチン化されたuS10を認識する(赤丸で表示)。RQTのSlh1ヘリカーゼサブユニットは、mRNAに引っ張り力を加え、主要なリボソームをサブユニットに解離させる。RQT 複合体によってmRNAが引っ張られた後、Cue2は衝突するリボソームから部分的に放出されたmRNAを切断する。
詳しくは、東京大学医科学研究所 のホームページをご覧ください。


